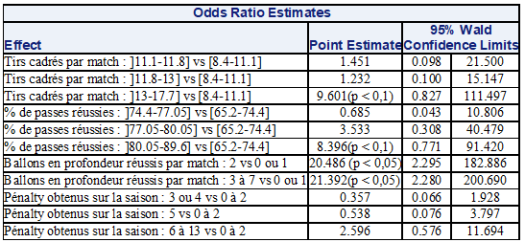
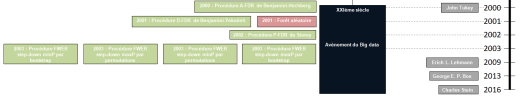
Présentation et démarche
Présentation et démarche
On présente ici deux analyses : celles des données du recensement de la population de 2012 puis de l’indice des prix mensuellement actualisé. Les variables sont directement récupérables sur le site de l’Insee. Pour le premier volet, on se restreindra à une approche commune en attendant une remise à plat du projet en fin d’année, qui s’orientera également vers une approche temporelle grâce aux données de 2017 publiées entre temps. De même pour le second volet, s’arrêtant à 2017 alors que les indicateurs sont publiés en continue jusqu’à aujourd’hui.
Un mot essentiel, la remise en question des données de l’Insee en France est assez coutumière, mais il est vrai qu’à Mayotte cela dépasse clairement l’entendement. L’Insee a pris le temps de publier sous format blog une discussion particulièrement intéressante de chaque argument sur lesquels elle est remise en question, malheureusement on entend encore des nombreux hauts placés les remettre en question. En fouillant un peu, en cherchant à être complémentaire au travail déjà produit et en imaginant que le débat soit fondé, on est alors heureux d’apprendre qu’à Mayotte, d’après ces hauts placés, il n’y a pas de sur-fécondité, pas de sur-mortalité, par de sur-recours aux pathologies infectieuses et encore plus fort, pas de sur-délinquance, etc. bref qu’à part un déficit de volume d’effectifs, déjà démontré avec les données de l’Insee, finalement tout va bien. Car « oui », dire que l’on est le double de ce que l’Insee avance, c’est diviser par deux fois plus chaque indicateur, l’exercice étant fait, voilà le constant que l’on en déduit. Cet effet est d’autant plus étonnant que les publications de l’Insee ne cesse de tirer la sonnette d’alarme sur la situation depuis plusieurs années, du point de vue de l’auteur de ce site, c’est une façon assez peu conventionnelle et extraordinaire de tirer sur l’un des défenseurs les plus pertinents et écoutés en France, mais bon comme l’a si bien dit son Directeur Général :
« On s’attaque au thermomètre plutôt qu’à la maladie »
Le raisonnement par l’absurde étant désormais fait, on partira du principe que les données de l’Insee sont fiables et que le projet proposé ici a un sens autant théorique que contextuel.
 Ahhhhhh… Mayotte
Ahhhhhh… Mayotte
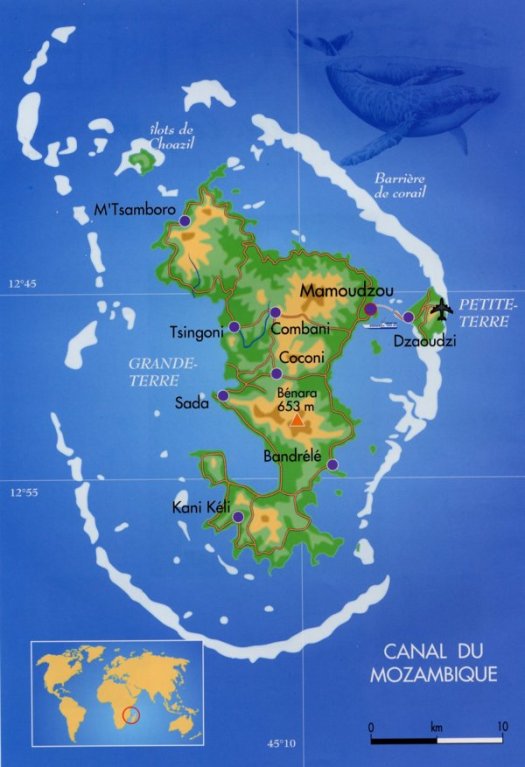
Le territoire de Mayotte est une île mesurant 376 km2 et se situe géographiquement dans l’archipel des Comores, lui-même situé dans l’océan Indien. Elle se divise en deux îles principales nommées la Grande-Terre et la Petite-Terre (oui bah l’une est plus petite que l’autre).
Historiquement, Mayotte était un territoire autonome jusqu’en 1841, date à laquelle le sultan prend la décision de vendre l’île à la France du fait de la pression des territoires voisins. Mayotte devient ainsi française en 1848. L’île, qui n’a jamais appartenu aux Comores donc, maintient ce premier statut suite à un vote ultra-majoritaire en 1976. Toujours sur la demande de sa population, elle vote majoritairement pour devenir un département d’Outre-mer français en 2009 et accède au statut de zone ultrapériphérique de l’Union Européenne en 2014. Remettre en question l’appartenance de Mayotte à la France, peut revenir d’une certaine manière à remettre en question celle de ma bien-aimée Corse ou même, pourquoi pas, de l’Alsace et la Lorraine, après tous les Mahorais sont français depuis plus longtemps que certains territoires rattachés à l’Hexagone directement…
Le territoire de divise en 17 communes, se déclinant en 72 villages. La commune de Mamoudzou rassemblant la plus grande partie de la population de Mayotte, elle figure comme la capitale économique de l’île.
Synthétiquement sur le plan socio-démographique, on rappellera qu’il s’agit du département le plus jeune de France avec un habitant sur deux âgé de 17 ans ou moins. Environ la moitié des habitants est de nationalité étrangère, dont la moitié en situation irrégulière, représentant un volume de tout de même près de 100 000 personnes alimentant ainsi le folklore local que l’on connait. Le contexte global tourne autour de la précarité avec quatre logements sur dix en tôle, trois sur dix n’ont pas accès à l’eau courante et les trois quarts de la population vivant en deçà du seuil de bas revenus métropolitains. L’Illettrisme concerne un habitant sur trois, seulement une personne sur quatre de 15 ans ou plus sort du système scolaire avec un diplôme qualifiant. Enfin, le secteur tertiaire est le plus représenté sur le territoire (concerne huit habitants sur dix) et plus précisément un habitant sur deux travaille dans la fonction publique.
Toujours le plus synthétiquement possible et sur le plan sanitaire cette fois-ci, le territoire est sujet à de nombreuses épidémies chaque année, à minima une par an depuis quelques temps. A cela s’ajoute un fort déficit en professionnels de santé et donc défaut de prise en charge, expliquant (de par bien d’autres indicateurs) qu’une personne née à Mayotte a globalement dix années de vie en moins qu’en France Hexagonale. Les données sur l’environnement sont assez faiblardes et on serait curieux de voir ce qu’elles pourraient offrir au diagnostic en Santé de Mayotte. En terme de pathologie, le diabète est particulièrement présent, tout comme les maladies infectieuses. On s’arrêtera là, très sincèrement, sur le plan Santé, tout est plus haut qu’en il ne le faut pas et plus bas quand il ne le faut pas qu’ailleurs.
Enfin, sur le plan sécuritaire, qui touche terriblement ce bout de territoire qui, il y a encore une vingtaine d’années était l’un des derniers paradis de la surface du globe provoquant un véritable phénomène dépressif à l’idée de le quitter, depuis 2011 la situation ne cesse de s’empirer sur cet aspect. Vivre à Mayotte est tristement devenu pénible et l’on espère de tout cœur que les mouvements sociaux en cours permettront à l’île de partir sur une nouvelle dynamique positive car, réellement, ce département est magnifique doublée d’une richesse culturelle incroyable et mériterait d’être un développement à l’aune de toutes ses qualités.
On pourra retrouver les éléments plus complets ayant alimenté ce micro-diagnostic subjectif de Mayotte ici :
https://www.mayotte.ars.sante.fr/le-panorama-sante-de-lars-mayotte
, le document reprenant en plus tout le volet bibliographique présenté initialement ici et supprimé depuis. A compléter avec la page wikipedia :
https://fr.wikipedia.org/wiki/Mayotte
 Exploitation des données du recensement de la population de 2012
Exploitation des données du recensement de la population de 2012
Les données : https://www.insee.fr/fr/statistiques/2409395?sommaire=2409812
Les données utilisées sont celles du recensement de la population de 2012 à l’échelle de la commune et sont donc déclaratives. Une base de données de 118 variables peut être construite, que l’on ramène à 86 variables optimisées en fonction de la taille de l’effectif ventilé sur les variables d’origines. Étant donné que l’on se lance dans une étude typologique, avec pour objectif de montrer la ressemblance entre les différentes communes, on a considéré la part en pourcentage de la ventilation des différents champs au sein des différentes thématiques pour chaque commune prise individuellement (en somme, une ligne équivaut à une commune). En effet, considérer les effectifs bruts ou bien la part en pourcentage en fonction des totaux mène à des résultats biaisés par le fait qu’il est évident que plus une commune concentre d’individus et de ménages et plus ses effectifs seront grands.
Deux types de données sont regroupées, celles issues du bulletin individuel et celles issues de la feuille logement.
- Pour les premières on trouve :
– La répartition par classes d’âge en sept catégories (0-4 ans, 5-19 ans, 20-29 ans, 30-39 ans, 40-49 ans, 50-64 ans et 65 ans ou plus) ;
– La répartition par genre ;
– La répartition par nationalité (français, étranger) ;
– La répartition par lieu de naissance (né à Mayotte, né ailleurs en France, né à l’Étranger) ;
– La répartition selon le statut matrimonial (célibataire-veuf-divorcé, marié) chez les 15 ans ou plus ;
– La répartition en fonction d’être en couple ou non chez les 15 ans ou plus ;
– La répartition en fonction du logement antérieur à Mayotte ou non ;
– La répartition selon la Catégorie Socio-professionnelle (CSP), au sens du recensement et non du Bureau International du Travail (BIT), renseignée chez les 15 ans ou plus (a un travail, chômeur, étudiants, retraité-au foyer-autres inactifs) ;
– La répartition selon le niveau de diplôme chez les 18 ans ou plus (sans scolarité, sans diplôme, CEP-BEPC-CAP-BEP, BAC-BAC pro, diplôme du premier cycle, diplôme du second ou troisième cycle).
- Et enfin, pour les données issues de la feuille de logement :
– La répartition par catégorie de logement (résidence principale, résidence vacante, logement secondaire-occasionnel) ;
– La répartition par aspect du bâti (habitation de fortune, habitation en dur, habitation traditionnelle-en bois) ;
– La répartition par type de construction pour les résidences principales (individuel, collectif) ;
– La répartition par statut d’occupation pour les résidences principales (propriétaire du logement uniquement, propriétaire du logement et du sol, locataire, logé gratuitement) ;
– La répartition par nature du sol pour les résidences principales (en terre battue, en béton, carrelage) ;
– La répartition par nombre de pièces pour les résidences principales (1 pièce, 2 pièces, 3-4 pièces, 5 pièces ou plus) ;
– La taille des ménages pour les résidences principales ;
– La répartition selon l’accessibilité par véhicule pour les résidences principales ;
– La répartition selon le nombre de voitures pour les résidences principales (aucune, une voiture, deux ou plus voitures) ;
– La répartition selon la présence ou non du confort de base pour les résidences principales (un logement est classé comme ayant le confort de base s’il a les WC, la baignoire, l’électricité et l’eau à l’intérieur du logement, si un seul de ces 4 items manque alors le logement est classé sans confort) ;
– La répartition selon le type d’accès à l’eau pour les résidences principales (eau froide à l’intérieur du logement seulement, eau froide et chaude à l’intérieur du logement, absence d’eau à l’intérieur et accès via une BFM, absence d’eau à l’intérieur et accès via un autre moyen qu’une BFM) ;
– La répartition selon la présence ou non de WC pour les résidences principales,
– La répartition selon la présence ou non d’électricité pour les résidences principales ;
– La répartition selon la présence ou non de baignoire pour les résidences principales ;
– La répartition selon le type d’évacuation des eaux usées pour les résidences principales (par le sol, par les égouts, par une fosse septique ;
– La répartition selon la présence ou non de bacs à ordure pour les résidences principales ;
– La répartition selon le mode de cuisson des aliments pour les résidences principales (cuisson par feu de bois, cuisson par réchaud à pétrole, gazinières, autres modes de cuisson) ;
– La répartition selon la présence ou non d’ordinateur pour les résidences principales ;
– La répartition selon la présence ou non de téléviseur pour les résidences principales ;
– La répartition selon la présence ou non de réfrigérateur pour les résidences principales ;
– La répartition selon la présence ou non de congélateur pour les résidences principales ;
– La répartition selon la présence ou non de lave-linge pour les résidences principales.
Cet inventaire est à garder à l’esprit pour les analyses à venir, car c’est à travers ce sous-ensemble de variables que l’on établira les différentes typologies. Évidemment, la considération d’autres caractéristiques amènerait à des résultats sensiblement différents, toutefois il est bon de savoir que l’on a là toutes les données socio-démographiques de base permettant d’ériger un distinguo « communes dans une situation globalement précaire » versus « communes plus développées ».
L’Analyse :
L’étude se déroulera en deux étapes :
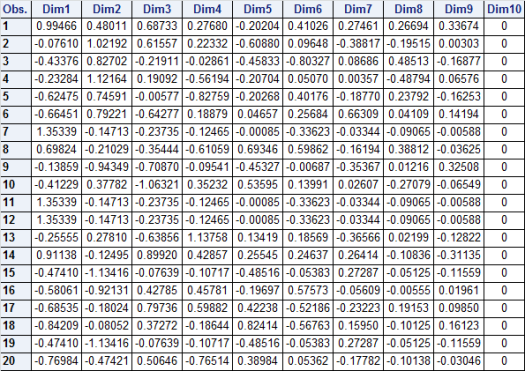
– La première étape consistera à procéder à une analyse exploratoire bivariée en faisant le point sur les corrélations entre les différentes variables au travers des coefficients de corrélation de Pearson, Spearman et Kendall puisque l’on a un jeu de données au format exclusivement continue et que de taille d’effectifs reste assez faible ( ) ;
) ;
– La seconde étape sera axée sur une étude typologique multivariée au travers d’une Analyse en Composantes Principales (ACP) et d’une Classification Ascendante Hiérarchique (CAH) afin de regrouper les communes en fonction des différents profils que l’on pourra mettre en évidence.
Donc on commence par la première phase des analyses. On lance sur l’ensemble des combinaisons, de 2 variables d’intérêt parmi les 86 variables présentes, le calcul des coefficients de corrélation cités ci-dessus, ce qui donne 3 655 combinaisons à éplucher. Le graphe ci-dessous présente le nombre de liaisons qui ressortent en fixant un 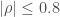 ,
,

En résumé sur l’ensemble des combinaisons et selon le seuil fixé, on a 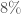 de corrélations significatives selon le coefficient de corrélation de Pearson,
de corrélations significatives selon le coefficient de corrélation de Pearson, 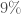 selon celui de Spearman et
selon celui de Spearman et  selon celui de Kendall. Face à autant de corrélations à parcourir, il convient de focaliser sur certaines variables importantes et partir du principe que l’on aura une vue plus exhaustive lors de l’approche multivariée. Ainsi, six thématiques sont jugées intéressantes, les croisements : avec l’âge, le genre, l’accès à l’eau, la CSP, la nationalité et le confort des logements.
selon celui de Kendall. Face à autant de corrélations à parcourir, il convient de focaliser sur certaines variables importantes et partir du principe que l’on aura une vue plus exhaustive lors de l’approche multivariée. Ainsi, six thématiques sont jugées intéressantes, les croisements : avec l’âge, le genre, l’accès à l’eau, la CSP, la nationalité et le confort des logements.
Pour les corrélations avec l’âge, quatre classes d’âges ressortent dans l’analyse bivariée. Les quatre figures qui suivent présentent les interactions avec la valeur du coefficient entre parenthèse et le sens de la corrélation (flèche) :
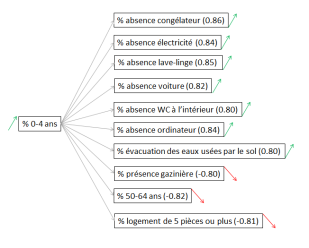
Globalement, la part des 0-4 ans croît avec celles du manque d’équipement et de la taille du logement. Assez logiquement, plus la part d’enfants en bas âge augmente et plus la part des personnes âgées diminue.
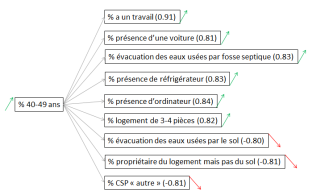
Globalement, la part des 40-49 ans croît en même temps que celles des conditions de vie et notamment le fait de travailler.

Globalement, la part des 50-64 ans croît en même temps que celles des équipements du ménage, des diplômés du BAC ou d’un BAC professionnel et du nombre de français.

Globalement, la part des 65 ans ou plus croît en même temps que celles des français et de ceux ayant l’eau froide seulement à l’intérieur du logement. A contrario, les parts des modes de cuisson par réchaud à pétrole et des étrangers diminuent quand elle augmente.
Pour les corrélations avec le genre, aucun croisement ne ressort, à la différence de l’accès à l’eau pour la part de ménages ayant seulement l’eau froide à l’intérieur du logement et pour celle des ménages s’approvisionnant en eau par un autre moyen que les BFM. Les deux figures qui suivent présentent les interactions avec la valeur du coefficient entre parenthèse et le sens de la corrélation (flèche) :

Globalement, la part des ménages avec seulement l’eau froide à l’intérieur du ménage croît en même temps que la qualité et la taille du logement.
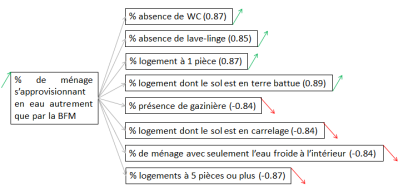
Globalement, la part des ménage s’approvisionnant en eau autrement que par la BFM croît en même temps que celles des logements précaires.
Pour les corrélations avec la CSP, deux catégories ressortent dans l’analyse bivariée, la part de ceux qui ont un travail et celle de ceux de CSP « autre » (retraité, au foyer, autres inactifs). Les trois figures qui suivent présentent les interactions avec la valeur du coefficient entre parenthèse et le sens de la corrélation (flèche) :

Globalement, la part de ceux qui ont un emploi croît avec celles des équipements du ménage ainsi que celle des 40-49 ans. De plus, elle est corrélée avec le niveau scolaire.

Globalement, la part de ceux ayant une CSP catégorisée « autre » croît avec l’absence d’équipements et de diplôme qualifiant. De plus, lorsque la part augmente, celles des 40-49 ans et 50-64 ans diminuent.
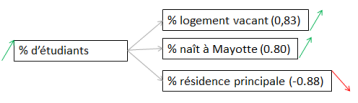
Globalement, la part d’étudiants croît en même temps que la part des catégories de logement et la part de nés à Mayotte.
Pour les corrélations avec la nationalité, les deux catégories ressortent dans l’analyse bivariée. Les deux figures qui suivent présentent les interactions avec la valeur du coefficient entre parenthèse et le sens de la corrélation (flèche) :
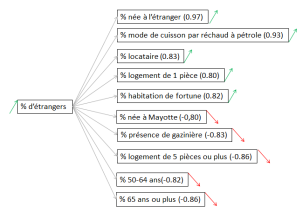
Globalement, la part des étrangers croît avec celles des logements précaires et de locataires. De plus, quand elle augmente, celles des 50-64 ans et des 65 ans ou plus diminuent.

Globalement, la part des français croît avec celles des logements de bonnes qualités. De plus, elle croît avec celles des 50-64 ans et des 65 ans ou plus.
Pour les corrélations avec le confort du logement, les deux catégories ressortent dans l’analyse bivariée. La figure qui suit présente les interactions avec la valeur du coefficient entre parenthèse et le sens de la corrélation (flèche) :

Globalement, l’indicateur du manque de confort reste assez robuste puisque, même s’il est basé sur uniquement quatre caractéristiques (WC à l’intérieur, baignoire, électricité, eau à l’intérieur), on voit que la part croît avec celles de l’absence de plusieurs autres équipements. De plus, quand elle augmente, celle des diplômés du BAC ou du BAC professionnel diminue. On notera que l’on ne présente pas l’analyse des corrélations avec la part des logements avec confort car elle conduit aux mêmes résultats inversés.
La seconde étape consiste donc à procéder à une analyse exploratoire multivariée au travers d’une ACP. Dans un premier temps, on détermine le nombre de composantes factorielles à retenir, on obtient alors le graphe de l’évolution des parts de variance suivant,

On constate que si on se limite aux deux premiers axes factoriels, c’est 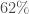 de la variance que l’on restitue. Pour une ACP cela semble un peu faible malgré le coude que l’on observe. Cependant, si on souhaite atteindre le gap classique de plus de
de la variance que l’on restitue. Pour une ACP cela semble un peu faible malgré le coude que l’on observe. Cependant, si on souhaite atteindre le gap classique de plus de 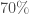 , on est obligé d’aller jusqu’à quatre axes factoriels, complexifiant un peu trop la lisibilité pour un gain plutôt limité. Afin de déterminer si on peut se limiter à deux composantes, on prend le temps de voir quelles oppositions chaque composante offre. On fixe le critère de significativité pour un coefficient sur la composante supérieur à
, on est obligé d’aller jusqu’à quatre axes factoriels, complexifiant un peu trop la lisibilité pour un gain plutôt limité. Afin de déterminer si on peut se limiter à deux composantes, on prend le temps de voir quelles oppositions chaque composante offre. On fixe le critère de significativité pour un coefficient sur la composante supérieur à 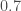 ou inférieur à
ou inférieur à 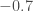 .
.
Pour la première composante, on constate l’opposition entre :
– La part des 40 à 64 ans, de ceux qui ont un emploi, de ceux ayant un BAC ou un BAC professionnel, de ceux ayant un diplôme du premier cycle, des logements en dur, des logements équipés d’un carrelage, des logements de trois pièces ou plus, des ménages avec une voiture, des logements catégorisés comme confortable, des logements équipés en WC, baignoire, électricité, ordinateur, téléviseur, réfrigérateur, congélateur, lave-linge et des logements dont l’évacuation des eaux usées se fait par fosse septique.
Soit des logements de bonnes qualités avec une population âgée qui travaille et est diplômée.
– Et la part des 0 à 4 ans, des CSP « autre » (retraité, au foyer, autres inactifs), des non scolarisés, des logements dont le sol est en terre, des logements de 1 à 2 pièce(s), des ménages sans voiture, des logements sans confort, des logements dont l’accès à l’eau se fait hors de la maison et pas par la BFM, des logements sans WC, sans électricité, sans baignoire, sans ordinateur, sans téléviseur, sans réfrigérateur, sans congélateur, sans lave-linge, dont l’évacuation des eaux usées se fait par le sol et dont le mode de cuisson se fait par le bois.
Soit des logements de mauvaise qualité, regroupant une grande part de jeunes enfants et d’une population en CSP « autre » et non scolarisée.
Pour la seconde composante, les oppositions sont déjà beaucoup moins nombreuses :
– La part des étrangers, de ceux nés à l’étranger, de ceux dont le logement antérieur n’était pas en France, de ceux ayant un diplôme du second ou troisième cycle, des locataires, de ceux ayant l’eau froide et chaude à l’intérieur du logement.
Soit une population étrangère diplômées et ayant un niveau de vie supérieur à la moyenne.
– Et la part des français, nés à Mayotte, dont le logement antérieur était à Mayotte, des étudiants et des logements vacants. Soit la population jeune et la population mahoraise.
Pour la troisième composante, on ne compte plus qu’une seule opposition significative : les logements collectifs versus les logement individuelles. Et pour la quatrième composante, aucune.
Ce premier bilan permet d’argumenter le choix de se restreindre à uniquement deux composantes étant donné que les composantes 3 et 4 n’apportent que peu d’information.
La figure ci-dessous présente la projection des variables sur les deux composantes retenues. En haut la projection de base et en bas le travail d’interprétation que l’on ajoute sur la figure de base :
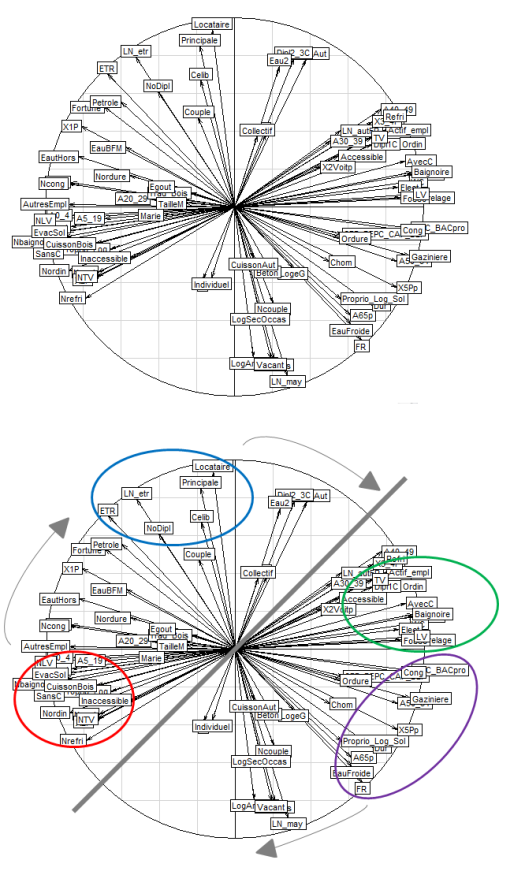
Quatre groupes principaux et trois groupes de transitions se dégagent. L’ACP permet d’ériger une carte caractéristique du type d’habitat, ainsi on a en rouge des logements précaires, en bleu ceux des étrangers, en vert des logement plus confortable et en violet les logements moins précaires.
– En rouge (s’oppose directement au groupe vert) : la population des 0-4 ans, les non scolarisés, les logements de 2 pièces, les ménages sans voiture, les logements sans confort, sans électricité, sans baignoire, sans ordinateur, sans téléviseur, sans réfrigérateur, sans lave-linge, inaccessible pour les voitures, les logements dont l’évacuation des eaux usées se fait par le sol, les ménages ayant recours à un mode de cuisson par bois.
– Le premier groupe de transition en gris, situé au dessus du groupe rouge: les ménages ayant recours aux réchauds à pétrole, les logements de fortune, les logements d’une pièce, les ménages sans eau à l’intérieur du logement et n’allant pas à la BFM s’approvisionner, les logements sans congélateur et les logements dont le sol est en terre battue.
– En bleu (s’oppose directement au groupe violet) : les locataires, les résidences principales, les nés à l’étranger, les célibataires (ou veufs ou divorcés), les étrangers et les sans diplôme.
– Le second groupe de transition en gris, situé à droite du groupe bleu : les logements ayant l’eau froide et l’eau chaude à l’intérieur du logement, les diplômés du second et troisième cycle et ceux dont le logement antérieur n’était pas en France.
– En vert (s’oppose directement au groupe rouge) : les logements équipés de réfrigérateur, les 40-49 ans, les logements de 3-4 pièces, ceux qui travaillent, les logements équipés de téléviseur, d’ordinateur, de baignoire, d’électricité, de lave-linge, de WC, les diplômés du premier cycle, les logements avec confort, les ménages ayant une voiture, les logements dont le sol est en carrelage et les logements dont l’évacuation des eaux usées se fait par fosse septique.
– En violet (s’oppose directement avec le groupe bleu) : les diplômés d’un BAC ou d’un BAC professionnel, les logements avec congélateur, gazinière, les 50 ans ou plus, les logements de 5 pièces ou plus, les propriétaires du logement et du sol, les habitations en dur, les logements avec uniquement l’eau froide à l’intérieur et les français.
– Et le troisième groupe de transition en gris, et situé en bas du groupe violet : les nés à Mayotte, les résidences vacantes, ceux dont le logement antérieur était à Mayotte et les étudiants.
On a donc une première typologie avec le groupe rouge représentant la population des très précaires. Ensuite le groupe bleu, qui est indépendant au groupe rouge et au groupe vert, se situe à côté des deux groupes gris de transition (gauche, précarité, et haut, développé) et qui s’inscrit finalement dans une classe intermédiaire. Puis le groupe vert qui, en opposition directe avec le groupe rouge, indique une population plus riche et qui reste très proche du groupe violet qui présente une forte corrélation en fonction de la tranche d’âge et du nombre de français. Puis le dernier groupe de transition, désignant la population des mahorais, étudiants, dont l’opposition se fait directement avec le groupe bleu des étrangers. A noter que les trois groupes de transition en gris sont indépendants les uns des autres, soit précarité, développé et jeunesse non liés.
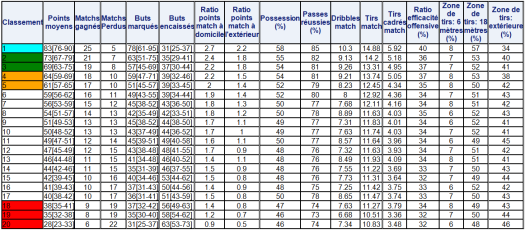
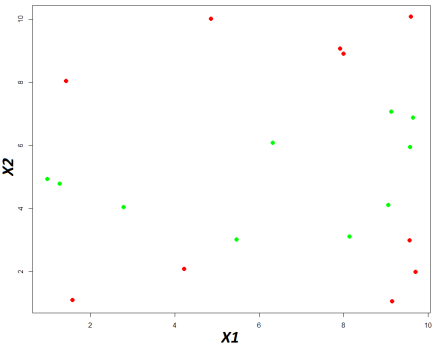
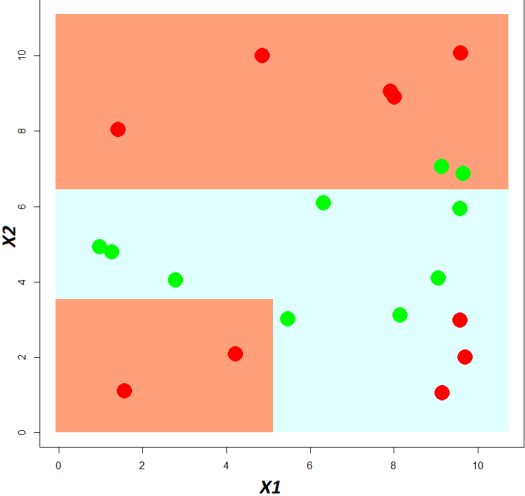
On va boucler l’étude en focalisant sur la ressemblance entre les communes au travers de la caractérisation obtenue par l’ACP. La figure suivante présente, à droite, la projection des observations soit des communes,

Afin d’objectiver le regroupement des communes, une classification ascendante hiérarchique est réalisée,

Quatre profils de communes se dégagent :
– Pamandzi, Dzadouzi et Sada, dont les projections coïncident avec la typologie des zones développées ;
– Boueni et Kani-Kéli, dont les projections coïncident avec l’autre typologie des zones développées;
– Chiconi, Mtsangamouiji, Acoua et Mtsamboro dont les projections coïncident avec la typologie des jeunes ;
– Mamoudzou, Chirongui, Dandrélé, Tsingoni, dont les projections coïncident avec la typologie des étrangers et des zones précaires ;
– Bandraboua, Koungou, Dembeni et Ouangani, dont la projection coïncident avec la typologie des zones précaires.
Et l’on finalise tout cela par un résultat visuel et simple à lire. On rappelle que l’on est sur les données de 2012 :

 Analyse spatiale de l’indice des prix
Analyse spatiale de l’indice des prix
Parmi les nombreux jeux de données récupérables depuis le site de l’Insee, il en est un particulièrement intéressant : l’indice des prix à la consommation. On dispose ainsi d’une série chronologique allant de 1999 à 2017. N’étant pas expert sur le sujet, il semble que les données sont réajustées en fonction d’une année de référence, nommée base, faisant qu’il est difficile de travailler directement sur l’ensemble de toute la série. Néanmoins, le site de l’Insee permet d’avoir une série actualisée débutant à janvier 2012 et disponible depuis cette page web (cf données complémentaires) :
– Série de données : https://www.insee.fr/fr/statistiques?debut=0&theme=80&categorie=2&geo=DEP-976
A toute fin utile, on rajoutera la page définissant les données utilisées ici : https://www.insee.fr/fr/metadonnees/definition/c1557
L’étude d’une telle série est l’occasion de se servir des outils d’analyse liés aux séries temporelles et aux chaînes de Markov. Mais avant cela, il est d’usage de décrire cette série de données au travers d’une analyse descriptive simple.

On a donc l’indice global des prix (bleu), celui lié à l’Alimentation (rouge), aux Services (orange), à l’Énergie (violet) et aux Produits Manufacturés (vert).
De manière général, l’indice des prix global a connu une forte hausse en 2012 qui se poursuit sur les cinq années suivantes mais de manière plus lente. Si l’on se réfère au graphe, les indices des prix de l’Alimentation et des Services augmentent depuis 2012, le premier nettement plus fortement que le second. A contrario, celui des Produits Manufacturés ne fait que chuter. Concernant celui des Énergies, il a connu une lente chute de 2012 à fin 2014 puis oscille jusqu’en mi-2017 pour finalement reprendre sa croissance en fin 2017. On s’attend à l’avenir à ce que l’indice des prix reprenne sa progression au vu des quatre autres courbes.
Si l’on raisonne en termes de taux de croissance mensuels, les éléments ci-dessous permettent une description plus précise du phénomène d’année en année :

A l’exception de l’année 2015 (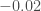 ), la croissance moyenne par mois est positive pour l’indice global des prix. Si elle reste relativement faible pour les années 2013 (
), la croissance moyenne par mois est positive pour l’indice global des prix. Si elle reste relativement faible pour les années 2013 (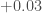 ), 2014 (
), 2014 (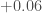 ), 2016 (
), 2016 (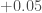 ) et 2017 (
) et 2017 (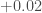 ), elle semble avoir connu un essor particulièrement important en 2012 (
), elle semble avoir connu un essor particulièrement important en 2012 ( ). Ce dernier constat est à relativiser étant donné la forte variabilité décrite par le calcul de l’écart-type,
). Ce dernier constat est à relativiser étant donné la forte variabilité décrite par le calcul de l’écart-type, 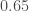 en 2012 versus
en 2012 versus 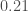 (2017),
(2017), 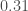 (2013, 2016),
(2013, 2016), 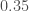 (2015).
(2015).
L’analyse des dotplots permet une caractérisation visuelle de la variabilité des taux de croissance mensuels pour chacune des sept années considérées :
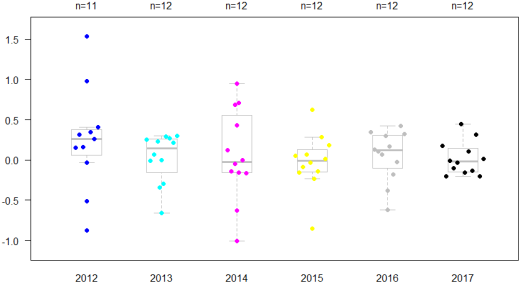
Cette approche met en évidence la grande variabilité évoquée des coefficients pour les années 2012 et 2014. Ce sont les années 2015 et 2017 qui, finalement, ont les plus faibles variabilités. Les années 2013 et 2016 montrant une tendance globale vers des taux de croissance mensuels négatifs à l’inverse de l’année 2017 dont la tendance est plutôt positive.
On peut également passer par une approche en trimestre en posant  la période de janvier, février, mars ;
la période de janvier, février, mars ; 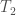 la période d’avril, mai, juin ;
la période d’avril, mai, juin ;  celle de juillet, août, septembre et
celle de juillet, août, septembre et 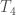 pour octobre, novembre, décembre :
pour octobre, novembre, décembre :

Il en ressort que les période avec les plus grandes variables sont le début de l’année ( ) avec une tendance globale positive et le début du second semestre (
) avec une tendance globale positive et le début du second semestre ( ) nulle.
) nulle. 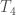 étant particulièrement compact en termes de distribution tout comme
étant particulièrement compact en termes de distribution tout comme 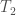 .
.
- Approche par chaînes de Markov
On peut considérer une modélisation de l’indice des prix global par les chaînes de Markov. Via cette approche, il ne sera pas possible de dépasser l’ordre trois étant donné qu’au-delà le nombre de cas devient trop faible pour établir des trajectoires suffisamment robustes.
En codant 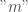 le cas où le coefficient de variation est inférieur à
le cas où le coefficient de variation est inférieur à 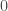 et
et 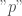 celui où il est supérieur à
celui où il est supérieur à 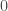 , on obtient les trois tableaux de trajectoires suivants avec en ligne les états précédents de la chaîne et en colonne l’état actuel :
, on obtient les trois tableaux de trajectoires suivants avec en ligne les états précédents de la chaîne et en colonne l’état actuel :

En prenant soin de considérer le rapport entre pourcentage de trajectoires et nombre de cas, on peut retenir les situations suivantes :
– pour l’ordre un, on a 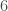 chances sur
chances sur 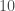 d’observer un coefficient positif après un coefficient négatif (trajectoire :
d’observer un coefficient positif après un coefficient négatif (trajectoire : 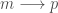 ).
).
– pour l’ordre deux,
 on a
on a 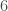 chances sur
chances sur 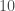 d’observer un coefficient positif après deux coefficients négatifs (trajectoire:
d’observer un coefficient positif après deux coefficients négatifs (trajectoire: 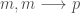 ) ;
) ;
 on a
on a 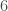 chances sur
chances sur 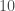 d’observer un coefficient positif après un coefficient négatif suivi d’un coefficient positif (trajectoire :
d’observer un coefficient positif après un coefficient négatif suivi d’un coefficient positif (trajectoire : 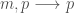 ) ;
) ;
 on a
on a 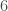 chances sur
chances sur 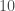 d’observer un coefficient positif après un coefficient positif suivi d’un coefficient négatif (trajectoire :
d’observer un coefficient positif après un coefficient positif suivi d’un coefficient négatif (trajectoire : 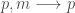 ).
).
– pour l’ordre trois,
 on a
on a  chances sur
chances sur 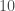 d’observer un coefficient positif après trois coefficients négatifs (trajectoire:
d’observer un coefficient positif après trois coefficients négatifs (trajectoire:  ) ;
) ;
 on a
on a  chances sur
chances sur 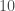 d’observer un coefficient positif après deux coefficients négatifs suivis d’un coefficient positif (trajectoire:
d’observer un coefficient positif après deux coefficients négatifs suivis d’un coefficient positif (trajectoire: 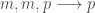 ) ;
) ;
 on a
on a 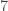 chances sur
chances sur 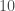 d’observer un coefficient positif après deux coefficients positifs suivis d’un coefficient négatif (trajectoire:
d’observer un coefficient positif après deux coefficients positifs suivis d’un coefficient négatif (trajectoire: 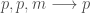 ) ;
) ;
 on a
on a 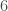 chances sur
chances sur 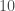 d’observer un coefficient négatifs après trois coefficients positifs (trajectoire:
d’observer un coefficient négatifs après trois coefficients positifs (trajectoire: 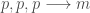 ).
).
Sur les huit cas que l’on a retenu, finalement deux sont inutiles (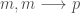 et
et 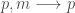 ) puisqu’ils présentent des probabilités similaires avec la trajectoire d’ordre un
) puisqu’ils présentent des probabilités similaires avec la trajectoire d’ordre un 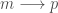 ).
).
En termes de prédiction, on est pour le mois de décembre 2017 sur un coefficient de variation positif mais on n’a pas pu déterminer de trajectoire suffisamment fiable pour rester sur l’ordre un. Si on se reporte à l’ordre deux, le mois de novembre 2017 est sur un coefficient de variation négatif ce qui renvoie au second cas de figure qui prédirait un coefficient positif en janvier 2018 dans 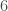 cas sur
cas sur 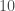 . En jetant un œil à l’ordre trois, le mois d’octobre 2017 est également associé à un coefficient de variation négatif, ce qui prédit également un coefficient positif dans
. En jetant un œil à l’ordre trois, le mois d’octobre 2017 est également associé à un coefficient de variation négatif, ce qui prédit également un coefficient positif dans 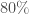 des cas. En consultant les données de l’Insee, on conste cependant que l’indice passe de
des cas. En consultant les données de l’Insee, on conste cependant que l’indice passe de  à
à 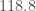 , soit une baisse, venant contredire cette prédiction.
, soit une baisse, venant contredire cette prédiction.
- Approche par série chronologique
L’échec de l’approche par chaîne de Markov est à modérer car les trajectoires retenues laissaient une grande part d’incertitude si l’on prend un peu de recul. On se propose cette fois-ci de passer par une tout autre approche, celle par série chronologique.
On a donc deux points à analyser avant de pouvoir se lancer dans les prédictions : la stationnarité et la saisonnalité. C’est deux points permettront de déterminer quel type de modèle l’on devra privilégier parmi les suivants : 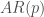 ,
, 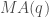 ,
, 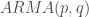 ,
,  ou
ou  .
.
L’étude des l’autocorrélogrammes est une étape importante dans l’analyse d’une série chronologique. Ci-dessous ceux obtenus sur les données :

On retrouve en haut à gauche la série de données d’origine et à sa droite l’autocorrélogramme. Ce dernier permet de tirer les cinq conclusions suivantes :
– Une décroissance lente des autocorrélations qui implique l’absence de stationnarité ;
– L’absence d’effet « sinusoïdale » des autocorrélations impliquant qu’il n’y a pas de saisonnalité ;
– L’aspect inversé qui dénote une inversion de tendance autour de la 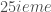 observation (soit janvier 2014) et qui peut s’expliquer par une saisonnalité observable de 2012 à 2013, tous les six mois, mais qui disparait pour les années 2014 à 2017 ;
observation (soit janvier 2014) et qui peut s’expliquer par une saisonnalité observable de 2012 à 2013, tous les six mois, mais qui disparait pour les années 2014 à 2017 ;
– Des autocorrélations qui, passées un retard de 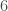 , deviennent non significatives ;
, deviennent non significatives ;
– Les autocorrélogrammes partiels (figure en bas à gauche) et inversés (en bas à droite) aurait tendance à s’orienter vers les paramètres 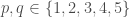 étant donné que pour ces lags, pas ou retard les autocorrélations restent significatives.
étant donné que pour ces lags, pas ou retard les autocorrélations restent significatives.
Cette première analyse permet d’exclure l’usage du modèle SARIMA qui est applicable aux séries présentant une saisonnalité et le modèle ARMA, quant à lui applicable à celles stationnaires . On s’orientera donc vers un modèle ARIMA.
Pour le choix du modèle optimal et donc des paramètres 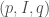 on va se baser sur les tests statistiques de Dickey-Fuller augmenté. Il faudra générer un nombre de lags, pas ou retards et fouiller dans les résultats des tests celui qui rejette l’hypothèse
on va se baser sur les tests statistiques de Dickey-Fuller augmenté. Il faudra générer un nombre de lags, pas ou retards et fouiller dans les résultats des tests celui qui rejette l’hypothèse  de racine unitaire.
de racine unitaire.
On va fixer un pas, lag ou retard de  à
à 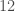 (en lien avec le nombre de mois sur une année) et observer le comportement des tests au fur et à mesure que l’on augmente le degré de différentiation
(en lien avec le nombre de mois sur une année) et observer le comportement des tests au fur et à mesure que l’on augmente le degré de différentiation 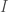 du modèle (et dont l’objectif est de rendre stationnaire le processus afin d’optimiser les prédictions). On obtient dés lors le tableaux de résultats suivants (le test « trend » pour l’étude de la tendance et le test single mean pour l’étude de l’écart à la moyenne globale):
du modèle (et dont l’objectif est de rendre stationnaire le processus afin d’optimiser les prédictions). On obtient dés lors le tableaux de résultats suivants (le test « trend » pour l’étude de la tendance et le test single mean pour l’étude de l’écart à la moyenne globale):

La condition pour retenir le meilleur modèle est le nombre de différentiation 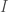 qui ne rejette pas
qui ne rejette pas 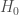 pour les quatre tests ci-dessus. Plusieurs combinaisons ressortent, il convient alors de retenir la plus simple soit celle présentant le moins de lags, pas ou retards. C’est le cas qui nécessite
pour les quatre tests ci-dessus. Plusieurs combinaisons ressortent, il convient alors de retenir la plus simple soit celle présentant le moins de lags, pas ou retards. C’est le cas qui nécessite 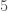 dérivations qui semblent répondre au mieux aux critères. Maintenant que l’on a déterminer le paramètre
dérivations qui semblent répondre au mieux aux critères. Maintenant que l’on a déterminer le paramètre 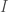 , il nous reste à déterminer ceux optimisant
, il nous reste à déterminer ceux optimisant 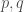 . Pour cela on teste les différentes combinaisons possibles dans l’ensemble que l’on a défini grâce à l’analyse des autocorrélogrammes et retenir celui qui minimise le critère
. Pour cela on teste les différentes combinaisons possibles dans l’ensemble que l’on a défini grâce à l’analyse des autocorrélogrammes et retenir celui qui minimise le critère 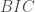 :
:

C’est donc le modèle ARIMA(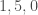 ) qui optimise la modélisation. Ce dernier étant établi, on passe maintenant aux prédictions. On génère alors le graphe suivant :
) qui optimise la modélisation. Ce dernier étant établi, on passe maintenant aux prédictions. On génère alors le graphe suivant :
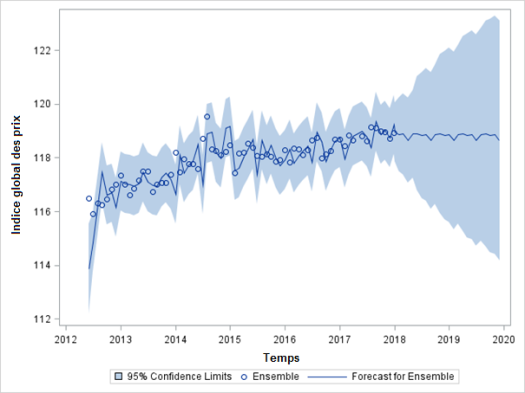
Pour l’année 2018, les prédictions envisagées de l’indice global des prix sont donc les suivantes :
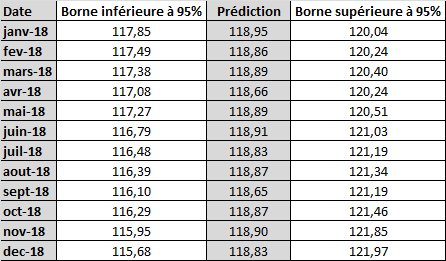
Si on compare avec les informations mises à disposition par l’Insee, il en ressort le constat suivant :
– Pour janvier, la valeur réelle de l’indice des prix global est de 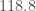 , le modèle ayant prédit une valeur très proche (
, le modèle ayant prédit une valeur très proche ( ) ;
) ;
– Pour février, la valeur réelle de l’indice des prix global est de  , le modèle ayant prédit une valeur peu éloignée (
, le modèle ayant prédit une valeur peu éloignée (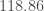 ) ;
) ;
– Pour mars, l’Insee n’a pas publié la valeur, aucun comparatif n’est possible ;
– Pour avril, la valeur réelle de l’indice des prix global est de 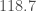 , le modèle ayant prédit une valeur très proche (
, le modèle ayant prédit une valeur très proche ( ) ;
) ;
En termes de tendance, le modèle avait prévu la chute de l’indice entre janvier et février (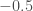 pour les valeurs réelles versus
pour les valeurs réelles versus 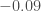 pour les valeurs prédites) mais avait également prédit une nouvelle chute pour avril alors que dans les faits l’indice global des prix est remonté (
pour les valeurs prédites) mais avait également prédit une nouvelle chute pour avril alors que dans les faits l’indice global des prix est remonté (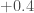 pour les valeurs réelles versus
pour les valeurs réelles versus  pour les valeurs prédites).
pour les valeurs prédites).
- Approche par régression linéaire multiple
Afin de conclure l’analyse, on proposons d’étudier la relation entre l’indice des prix global et ceux associés à l’Alimentation, aux Produits Manufacturés, à l’Énergie et aux Services. L’idée étant d’observer l’influence de chacun de ces indices sur le global.
Étant donné la nature des données qui sont de format continue, le modèle linéaire semble le plus approprié. Reste à définir si on reste sur une régression linéaire multiple ou si on opte pour sa version PLS. Afin d’argumenter ce futur choix, on regarde les matrices des corrélations de Pearson, Spearman et Kendall :

Les conclusions que l’on en tire à la lecture sont :
– Une forte corrélation linéaire entre les indices des prix de l’Alimentation et des Services avec l’indice global (respectivement 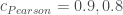 ). Ainsi, qu’une forte corrélation monotone décroissante avec les Produits Manufacturés (
). Ainsi, qu’une forte corrélation monotone décroissante avec les Produits Manufacturés (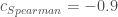 ). A contrario il n’y a pas de relation significative avec l’indice des prix associé à l’Énergie (
). A contrario il n’y a pas de relation significative avec l’indice des prix associé à l’Énergie (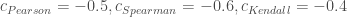 ).
).
– L’existence d’une forte multicolinéarité entre les indices des prix de l’Alimentation, des Produits Manufacturés, de l’Énergie et des services. Notamment la présence de deux groupes de variables avec, d’un côté, les indices associés à l’Alimentation et aux Services et, d’un autre côté, ceux de l’Énergie et des Produits Manufacturés.
Les corrélations et anti-corrélations sont particulièrement fortes, par conséquent on peut s’orienter vers un modèle linéaire PLS qui sera mieux adapté. La subtilité revient alors à déterminer le nombre de composantes PLS à retenir pour définir les coefficients de régression finaux. Pour cela, on étudie les performances du modèle en fonction de la variance expliquée pour les quatre composantes calculables :

On constate que la première composante explique 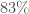 de la variance des quatre variables explicatives et
de la variance des quatre variables explicatives et 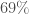 avec la variable à expliquer (Indice des prix global). En poussant le nombre de composantes, on parvient à des résultats très bons pour trois composantes,
avec la variable à expliquer (Indice des prix global). En poussant le nombre de composantes, on parvient à des résultats très bons pour trois composantes,  pour les deux. Par conséquent, le choix de trois composantes peut être retenu, ce qui donne les coefficients de régression linéaire PLS suivants :
pour les deux. Par conséquent, le choix de trois composantes peut être retenu, ce qui donne les coefficients de régression linéaire PLS suivants :

La sortie des coefficients permet de déterminer le poids des différents indices ainsi que leur influence sur l’indice des prix global. Ainsi, ce sont les indices liés à l’Alimentation et aux Produits Manufacturés qui pèsent le plus dans la croissance ou la décroissance de l’indice des prix global. Et si on se reporte aux coefficients standardisés, on en conclut que l’indice des prix de l’Alimentation a un impact 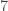 fois plus important que celui de l’Énergie et
fois plus important que celui de l’Énergie et 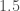 fois plus important que celui des Services. Quant à celui des Produits Manufacturés, le rapport de force est de
fois plus important que celui des Services. Quant à celui des Produits Manufacturés, le rapport de force est de 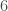 fois plus important par rapport aux Énergies et
fois plus important par rapport aux Énergies et  fois plus important pour les Services. C’est donc le rapport de force entre Alimentation et Produits Manufacturés qui sera déterminant, sachant qu’il tourne en faveur du premier avec un coefficient de
fois plus important pour les Services. C’est donc le rapport de force entre Alimentation et Produits Manufacturés qui sera déterminant, sachant qu’il tourne en faveur du premier avec un coefficient de  fois plus impactant que le second.
fois plus impactant que le second.